EM画像を対象にした粒子のインスタンスセグメンテーションモデルが下記論文で報告されていたので手持ちの画像で試してみました。
Bayesian Particle Instance Segmentation for Electron Microscopy Image QuantificationBatuhan Yildirim and Jacqueline M. ColeJournal of Chemical Information and Modeling 2021 61 (3), 1136-1149DOI: 10.1021/acs.jcim.0c01455
提案モデルは、通常のインスタンスセグメンテーションモデルとは異なり、各画素が帰属する「粒子の重心座標」を予測するタスクに置き換えられている。これによってend-to-endでインスタンスセグメンテーションを実施できている(元ネタはこちらの論文:Instance Segmentation by Jointly Optimizing Spatial Embeddings and Clustering Bandwidth, arXiv:1906.11109)。
予測タスクを抽象化したことで、同じ重みのモデルで複数種類の材料画像に適用できるとのこと。
また、推論時にもドロップアウトを使用することで予測値を分布として取得し、その分散から予測の信頼性を評価して、誤検出を抑制している。
著者公開コード
著者らがgithubで公開しているコードは以下。
今回は上記コードをベースにして、使いやすいように一部修正して動かした。
ディレクトリ構成
ディレクトリ構成は以下の通り。コードの詳細はページ下部にまとめた。
.
└── bpartis
├── models
| └seg-model.pt
└── segment
├── model.py
├── nnmodules.py
├── cluster.py
├── uncertainty.py
└── visualization.py学習済み重み(seg-model.pt)は、以下を使用。
実際に動かしてみた
ライブラリをインポート。
import os
import numpy as np
import cv2
import matplotlib.pyplot as plt
from bpartis.segment.model import ParticleSegmenter
from bpartis.segment.visualization import vis_segmap
テスト用画像(test2imageモデルで生成した画像)を読み込む。この画像に映っている白濁粒子をきれいにセグメンテーションできるか試してみる。ちなみに、モデルはEM画像(グレースケール画像)で学習されているが、テストにはカラー画像を適用している。
image = cv2.imread('./test.png')
plt.imshow(cv2.cvtColor(image, cv2.COLOR_BGR2RGB))
plt.show()
モデルを読み込み、セグメンテーションを実施、その結果をインスタンスごとに塗り分けて表示した。その結果、きれいにセグメンテーションできていることが分かる。
segmenter = ParticleSegmenter(model_path='../models/seg-model.pt', device='cpu')
segmentation, uncertainty, _ = segmenter.segment(image)
os.makedirs('./results', exist_ok=True)
cv2.imwrite('./results/test_seg.png', segmentation)
seg_cl, seg_cl_concat = vis_segmap(segmentation, image=image)
fig = plt.figure(figsize=(10,10))
ax = fig.add_subplot(2,1,1)
ax.imshow(seg_cl)
ax = fig.add_subplot(2,1,2)
ax.imshow(seg_cl_concat)
plt.show()
モデルの出力の確認。入力画像と同じ形をしたndarrayで、画素値がインスタンスごとのidとなっている。
print('shape:',segmentation.shape)
print('labels:', np.unique(segmentation))shape: (512, 512)
labels: [ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23
24 25 26 27 28 29 30 31]
予測結果の分散から求めた、予測の不確かさを出力。粒子の領域は暗く(=不確かさが小さい)、粒子境界や、粒子が無い領域で明るく(=不確かさが大きい)なっていることが分かる。ベイズ推論の際は、この不確かさがしきい値以上の領域を除外するようにして誤検出を抑制している。
fig, ax = plt.subplots()
im = ax.imshow(uncertainty)
fig.colorbar(im, ax=ax)
plt.show()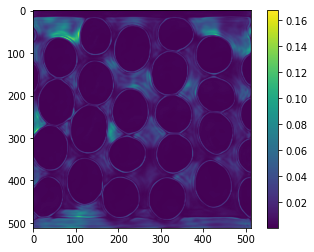
GPUを使用する場合は、モデル読み込みの際の引数にdevice=’cuda’を入れる。
segmenter = ParticleSegmenter(model_path='../models/seg-model.pt',device='cuda')
また、ベイズ推論する必要が無い場合は、モデル読み込みの際の引数にbayesian=Falseを入れる。ベイズ推論時はドロップアウトを有効にして複数回(デフォルトでは30回)のフォワード処理をしているが、bayesian=Falseにするとフォワード処理が1回のみになるので処理時間が短くなるメリットがある。一方で、予測結果の不確かさを考慮できないため、誤検出が増えるデメリットがある。
今回のテスト画像でも、粒子の無い領域に誤検出がみられる。
segmenter = ParticleSegmenter(bayesian=False, model_path='../models/seg-model.pt',device='cuda')
segmentation, uncertainty, _ = segmenter.segment(image)
fig = plt.figure(figsize=(10,10))
ax = fig.add_subplot(2,1,1)
ax.imshow(seg_cl)
ax = fig.add_subplot(2,1,2)
ax.imshow(seg_cl_concat)
plt.show()
コード詳細
githubはこちら
import os
import torch
import numpy as np
from PIL import Image
from .cluster import Cluster
from .nnmodules import BranchedERFNet
from .uncertainty import expected_entropy, predictive_entropy, uncertainty_filtering
# model_pathを指定できるように追記
class ParticleSegmenter:
def __init__(self, bayesian=True, n_samples=30, tu=0.0125, model_path='../models/seg-model.pt', device='cpu'):
self.bayesian = bayesian
self.n_samples = n_samples
self.tu = tu
self.seg_model = BranchedERFNet(num_classes=[4, 1]).to(device).eval()
self.model_path = os.path.join(os.path.dirname(os.path.abspath(__file__)), model_path)
self.seg_model.load_state_dict(torch.load(self.model_path, map_location=device))
self.cluster = Cluster(n_sigma=2, device=device)
if device is None:
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
self.device = device
def preprocess(self, image):
"""Pre-process image for segmentation model."""
image = Image.fromarray(image)
image = image.resize((512, 512), resample=Image.BICUBIC)
image = np.array(image)
image = image / 255.0
return image
def postprocess_pred(self, image, h, w):
"""Post-process output segmentation map. Return output to the original input size."""
image = Image.fromarray(image)
image = image.resize((w, h), resample=Image.NEAREST)
return np.array(image)
def postprocess_uncertainty(self, image, h, w):
"""
Resize uncertainty map. This is strictly for visualisation purposes.
The output of this function will not be used for anything other
than visualisation.
"""
image = Image.fromarray(image)
image = image.resize((w, h), resample=Image.BICUBIC)
return np.array(image)
def enable_eval_dropout(self):
"""Enables dropout in eval mode for Bayesian inference via Monte Carlo dropout."""
for module in self.seg_model.modules():
if 'Dropout' in type(module).__name__:
module.train()
def monte_carlo_predict(self, image):
"""Performs Bayesian inference and computes epistemic uncertainty."""
h, w = image.shape[-2:]
cluster = Cluster(n_sigma=2, h=h, w=w, device=self.device)
self.enable_eval_dropout()
# get monte carlo model samples
mc_outputs = []
mc_seed_maps = []
for i in range(self.n_samples):
output = self.seg_model(image).detach()
seed_map = torch.sigmoid(output[0, -1]).unsqueeze(0) # \phi_{k}(e_{i})
mc_outputs.append(output)
mc_seed_maps.append(seed_map)
mc_outputs = torch.cat(mc_outputs, dim=0)
mc_seed_maps = torch.cat(mc_seed_maps, dim=0)
# MC prediction (cluster the mean of MC samples)
mc_prediction, _ = cluster.cluster(mc_outputs.mean(dim=0))
# Uncertainty
total = predictive_entropy(mc_seed_maps)
aleatoric = expected_entropy(mc_seed_maps)
epistemic = total - aleatoric # $MI(y, \theta | x)$
return mc_prediction, epistemic
def segment(self, image):
"""Main segmentation routine."""
o_h, o_w = image.shape[:2]
image = self.preprocess(image)
image = torch.FloatTensor(image).permute(2, 0, 1).unsqueeze(0).to(self.device)
if self.bayesian:
# monte carlo predict
pred, uncertainty = self.monte_carlo_predict(image)
original = pred.cpu().numpy().copy()
original = self.postprocess_pred(original, o_h, o_w)
pred = uncertainty_filtering(pred, uncertainty, tu=self.tu)
pred = pred.cpu().numpy()
uncertainty = uncertainty.cpu().numpy()
# post-process uncertainty for visualisation
uncertainty = self.postprocess_uncertainty(uncertainty, o_h, o_w)
else:
model_out = self.seg_model(image)[0].detach()
pred = self.cluster.cluster(model_out)[0].cpu().numpy()
uncertainty = None
original = None
pred = self.postprocess_pred(pred, o_h, o_w)
return pred, uncertainty, originalimport torch
import torch.nn as nn
import torch.nn.functional as F
class DownsamplerBlock (nn.Module):
def __init__(self, ninput, noutput):
super().__init__()
self.conv = nn.Conv2d(ninput, noutput-ninput,
(3, 3), stride=2, padding=1, bias=True)
self.pool = nn.MaxPool2d(2, stride=2)
self.bn = nn.BatchNorm2d(noutput, eps=1e-3)
def forward(self, input):
output = torch.cat([self.conv(input), self.pool(input)], 1)
output = self.bn(output)
return F.elu(output)
class non_bottleneck_1d (nn.Module):
def __init__(self, chann, dropprob, dilated):
super().__init__()
self.conv3x1_1 = nn.Conv2d(
chann, chann, (3, 1), stride=1, padding=(1, 0), bias=True)
self.conv1x3_1 = nn.Conv2d(
chann, chann, (1, 3), stride=1, padding=(0, 1), bias=True)
self.bn1 = nn.BatchNorm2d(chann, eps=1e-03)
self.conv3x1_2 = nn.Conv2d(chann, chann, (3, 1), stride=1, padding=(
1*dilated, 0), bias=True, dilation=(dilated, 1))
self.conv1x3_2 = nn.Conv2d(chann, chann, (1, 3), stride=1, padding=(
0, 1*dilated), bias=True, dilation=(1, dilated))
self.bn2 = nn.BatchNorm2d(chann, eps=1e-03)
self.dropout = nn.Dropout2d(dropprob)
def forward(self, input):
output = self.conv3x1_1(input)
output = F.elu(output)
output = self.conv1x3_1(output)
output = self.bn1(output)
output = F.elu(output)
output = self.conv3x1_2(output)
output = F.elu(output)
output = self.conv1x3_2(output)
output = self.bn2(output)
if (self.dropout.p != 0):
output = self.dropout(output)
return F.elu(output+input)
class Encoder(nn.Module):
def __init__(self, num_classes):
super().__init__()
self.initial_block = DownsamplerBlock(3, 16)
self.layers = nn.ModuleList()
self.layers.append(DownsamplerBlock(16, 64))
for x in range(0, 5): # 5 times
self.layers.append(non_bottleneck_1d(64, 0.03, 1))
self.layers.append(DownsamplerBlock(64, 128))
for x in range(0, 2): # 2 times
self.layers.append(non_bottleneck_1d(128, 0.3, 2))
self.layers.append(non_bottleneck_1d(128, 0.3, 4))
self.layers.append(non_bottleneck_1d(128, 0.3, 8))
self.layers.append(non_bottleneck_1d(128, 0.3, 16))
# Only in encoder mode:
self.output_conv = nn.Conv2d(
128, num_classes, 1, stride=1, padding=0, bias=True)
def forward(self, input, predict=False):
output = self.initial_block(input)
for layer in self.layers:
output = layer(output)
if predict:
output = self.output_conv(output)
return output
class UpsamplerBlock (nn.Module):
def __init__(self, ninput, noutput):
super().__init__()
self.conv = nn.Conv2d(ninput, noutput, 3, stride=1, padding=1, bias=True)
self.bn = nn.BatchNorm2d(noutput, eps=1e-3)
def forward(self, input):
output = F.interpolate(input, scale_factor=2, mode='bilinear', align_corners=False)
output = self.conv(output)
output = self.bn(output)
return F.elu(output)
class Decoder (nn.Module):
def __init__(self, num_classes):
super().__init__()
self.layers = nn.ModuleList()
self.layers.append(UpsamplerBlock(128, 64))
self.layers.append(non_bottleneck_1d(64, 0, 1))
self.layers.append(non_bottleneck_1d(64, 0, 1))
self.layers.append(UpsamplerBlock(64, 16))
self.layers.append(non_bottleneck_1d(16, 0, 1))
self.layers.append(non_bottleneck_1d(16, 0, 1))
self.output_conv = nn.Conv2d(16, num_classes, 3, stride=1, padding=1, bias=True)
def forward(self, input):
output = input
for layer in self.layers:
output = layer(output)
output = F.interpolate(output, scale_factor=2, mode='bilinear', align_corners=False)
output = self.output_conv(output)
return output
# ERFNet
class Net(nn.Module):
def __init__(self, num_classes, encoder=None): # use encoder to pass pretrained encoder
super().__init__()
if (encoder == None):
self.encoder = Encoder(num_classes)
else:
self.encoder = encoder
self.decoder = Decoder(num_classes)
def forward(self, input, only_encode=False):
if only_encode:
return self.encoder.forward(input, predict=True)
else:
output = self.encoder(input) # predict=False by default
return self.decoder.forward(output)
class BranchedERFNet(nn.Module):
def __init__(self, num_classes, encoder=None):
super().__init__()
if (encoder is None):
self.encoder = Encoder(sum(num_classes))
else:
self.encoder = encoder
self.decoders = nn.ModuleList()
for n in num_classes:
self.decoders.append(Decoder(n))
def init_output(self, n_sigma=1):
with torch.no_grad():
output_conv = self.decoders[0].output_conv
print('initialize last layer with size: ',
output_conv.weight.size())
output_conv.weight[:, 0:2, :, :].fill_(0)
output_conv.bias[0:2].fill_(0)
output_conv.weight[:, 2:2+n_sigma, :, :].fill_(0)
output_conv.bias[2:2+n_sigma].fill_(1)
def forward(self, input, only_encode=False):
if only_encode:
return self.encoder.forward(input, predict=True)
else:
output = self.encoder(input)
return torch.cat([decoder.forward(output) for decoder in self.decoders], 1)import torch
class Cluster:
def __init__(self, n_sigma=2, h=512, w=512, device='cuda'):
self.n_sigma = n_sigma
self.device = device
xm = torch.linspace(0, 1, w).view(1, 1, -1).expand(1, h, w)
ym = torch.linspace(0, 1, h).view(1, -1, 1).expand(1, h, w)
xym = torch.cat((xm, ym), 0)
self.xym = xym.to(self.device)
def cluster(self, prediction, threshold=0.5):
height, width = prediction.size(1), prediction.size(2)
xym_s = self.xym[:, 0:height, 0:width]
spatial_emb = torch.tanh(prediction[0:2]) + xym_s # 2 x h x w
sigma = prediction[2:2+self.n_sigma] # n_sigma x h x w
seed_map = torch.sigmoid(prediction[2+self.n_sigma:2+self.n_sigma + 1]) # 1 x h x w
instance_map = torch.zeros(height, width).short()
instances = []
count = 1
mask = (seed_map > 0.5).bool()
if mask.sum() > 128:
spatial_emb_masked = spatial_emb[mask.expand_as(spatial_emb)].view(2, -1)
sigma_masked = sigma[mask.expand_as(sigma)].view(self.n_sigma, -1)
seed_map_masked = seed_map[mask].view(1, -1)
unclustered = torch.ones(mask.sum()).short().to(self.device)
instance_map_masked = torch.zeros(mask.sum()).short().to(self.device)
while(unclustered.sum() > 128):
seed = (seed_map_masked * unclustered.float()).argmax().item()
seed_score = (seed_map_masked * unclustered.float()).max().item()
if seed_score < threshold:
break
center = spatial_emb_masked[:, seed:seed+1]
unclustered[seed] = 0
s = torch.exp(sigma_masked[:, seed:seed+1]*10)
dist = torch.exp(-1*torch.sum(torch.pow(spatial_emb_masked -
center, 2)*s, 0, keepdim=True))
proposal = (dist > 0.5).squeeze()
if proposal.sum() > 128:
if unclustered[proposal].sum().float()/proposal.sum().float() > 0.5:
instance_map_masked[proposal.squeeze()] = count
instance_mask = torch.zeros(height, width).short()
instance_mask[mask.squeeze().cpu()] = proposal.cpu().short()
instances.append(
{'mask': instance_mask.squeeze()*255, 'score': seed_score})
count += 1
unclustered[proposal] = 0
instance_map[mask.squeeze().cpu()] = instance_map_masked.cpu()
return instance_map, instancesimport torch
def entropy(p, eps=1e-6):
p = torch.clamp(p, eps, 1.0-eps)
return -1.0*((p*torch.log(p)) + ((1.0-p)*(torch.log(1.0-p))))
def expected_entropy(mc_preds):
"""Aleatoric (data) uncertainty"""
return torch.mean(entropy(mc_preds), dim=0)
def predictive_entropy(mc_preds):
"""Total uncertainty"""
return entropy(torch.mean(mc_preds, dim=0))
def uncertainty_filtering(prediction, uncertainty, tu=0.0125):
"""Filters instance segmentaton predictions based on their uncertainty."""
filtered_pred = torch.zeros_like(prediction)
for inst_id in torch.unique(prediction):
if inst_id == 0:
continue
inst_mask = prediction == inst_id
inst_uncertainty = torch.mean(uncertainty[inst_mask])
if inst_uncertainty < tu:
filtered_pred[inst_mask] = torch.max(filtered_pred) + 1
return filtered_pred予測結果を描画するメソッドを新たに作成。
import numpy as np
import cv2
import matplotlib.pyplot as plt
def vis_segmap(segmentation, cm_name='gist_ncar', image=[]):
cm = plt.get_cmap(cm_name)
x,y = segmentation.shape
seg_cl = np.zeros((x,y,3))
for i in list(np.unique(segmentation)):
# 背景はスキップ
if i==0:
continue
cl_num = i/23
cl_num = cl_num - int(cl_num)
seg_cl[segmentation==i] = cm(cl_num)[:3]
if image==[]:
return seg_cl
else:
seg_cl_concat = (cv2.cvtColor(image, cv2.COLOR_BGR2RGB)/255 + seg_cl)*0.5
return seg_cl, seg_cl_concatまとめ
今回は、粒子画像のインスタンスセグメンテーションモデルであるBPartISを試した。未学習のテスト画像でもしっかりセグメンテーションできることが確認できた。

コメント