Rとstanを使って、正規分布から生成したデータの平均と標準偏差をMCMC法でベイズ推定しました。最近、こちらの本でベイズ統計の勉強中なので備忘録として投稿します。
Amazon.co.jp: 実践Data Scienceシリーズ RとStanではじめる ベイズ統計モデリングによるデータ分析入門 (KS情報科学専門書) eBook : 馬場真哉: Kindleストア
Amazon.co.jp: 実践Data Scienceシリーズ RとStanではじめる ベイズ統計モデリングによるデータ分析入門 (KS情報科学専門書) eBook : 馬場真哉: Kindleストア
データ
平均100、標準偏差10の正規分布からサンプルを100個生成します。
# 平均100、標準偏差が10の乱数(実数)を100個作成
set.seed(5)
d <- rnorm(100, mean = 100, sd = 10)ヒストグラムをプロットしてデータを確認します。
# ヒストグラム
png("plot1.png")
hist(d)
dev.off()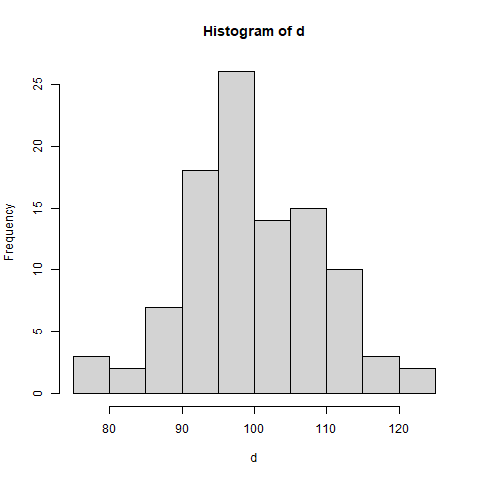
処理
MCMC用のstanファイルを作成します。
data {
/* ... declarations ... */
int N; //サンプルサイズ
vector[N] values; //データ
}
parameters {
/* ... declarations ... */
real mu; //平均
real<lower=0> sigma; //標準偏差
}
model {
/* ... declarations ... statements ... */
for (i in 1:N) {
values[i] ~ normal(mu, sigma);
}
}
データサイズの取得、データをリスト化したうえで、stanを実行します。
library(rstan)
# 計算の高速化
rstan_options(auto_write = TRUE)
options(mc.cores = parallel::detectCores())
# データ整形
sample_size <- length(d)
data_list <- list(values = d, N = sample_size)
# stan実行
mcmc_result <- stan(
file = "01_test.stan",
data = data_list,
seed = 1,
chains = 4,
iter = 3000,
warmup = 1000,
thin = 2
)
# 結果出力
print(mcmc_result)
# トレースプロット
png("plot2.png", 640, 400)
traceplot(mcmc_result)
dev.off()出力:
Inference for Stan model: test.
4 chains, each with iter=3000; warmup=1000; thin=2;
post-warmup draws per chain=1000, total post-warmup draws=4000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
mu 100.32 0.02 0.97 98.43 99.68 100.32 100.95 102.14 3313 1
sigma 9.57 0.01 0.69 8.33 9.10 9.53 10.01 11.04 3709 1
lp__ -272.90 0.02 1.03 -275.57 -273.29 -272.60 -272.17 -271.91 2985 1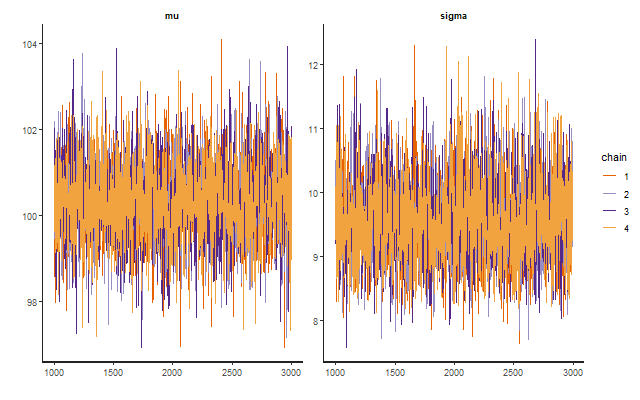
Rhatが1.1以下なのでちゃんと収束できたようです。トレースプロットからも定常分布に収束していることが分かります。推定値は、中央値で見ると平均100.32 標準偏差9.53、95%信頼区間で見ると[98.43,102.14]となっており、生成元の確率分布=N(100,10)に近い値を出せています。
他のプロット方法
トレースプロットをwarmup期間も含めてプロット
# トレースプロット、warmup含む
png("plot3.png", 640, 400)
traceplot(mcmc_result, inc_warmup=T)
dev.off()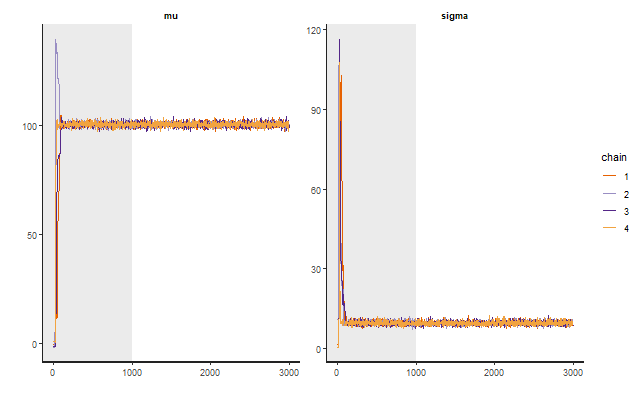
ggfortifyを使ってトレースプロット、平均μのみ
library(ggfortify)
# ggfortifyでトレースプロット
png("plot4.png", 800, 400)
autoplot(ts(mcmc_sample[,,"mu"]),
facets = F,
ylab = "mu",
main = "トレースプロット")
dev.off()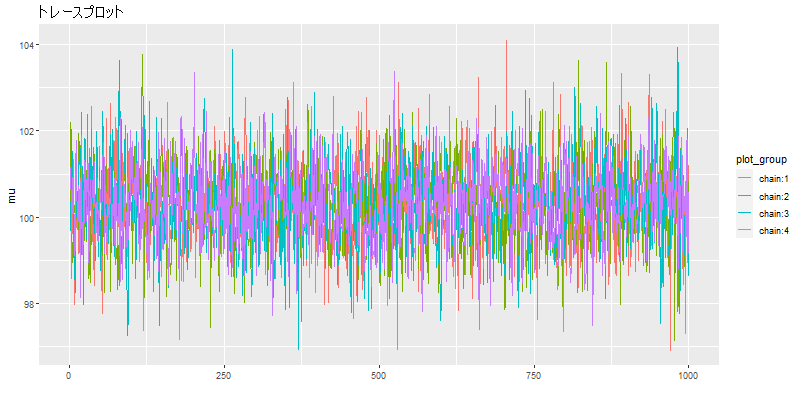
bayseplotを使って結果をプロット
library(bayesplot)
# MCMCサンプルのヒストグラム
png("plot5.png", 640, 400)
mcmc_hist(mcmc_sample, pars=c("mu","sigma"))
dev.off()
# 事後分布
png("plot6.png", 640, 400)
mcmc_dens(mcmc_sample, pars=c("mu","sigma"))
dev.off()
# トレースプロット
png("plot7.png", 640, 400)
mcmc_trace(mcmc_sample, pars=c("mu","sigma"))
dev.off()
# 事後分布とトレースプロットをまとめて図示
png("plot8.png", 640, 400)
mcmc_combo(mcmc_sample, pars=c("mu","sigma"))
dev.off()MCMCサンプルのヒストグラム
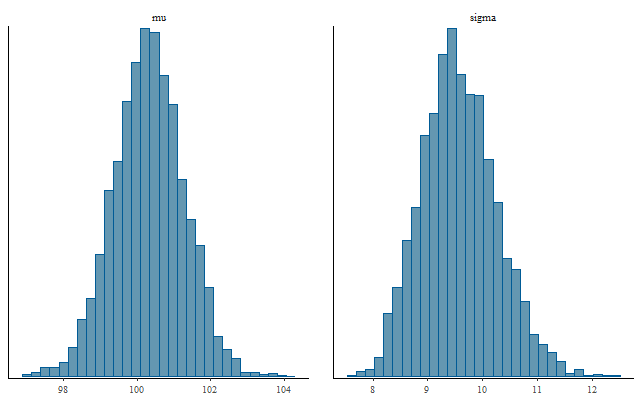
事後分布
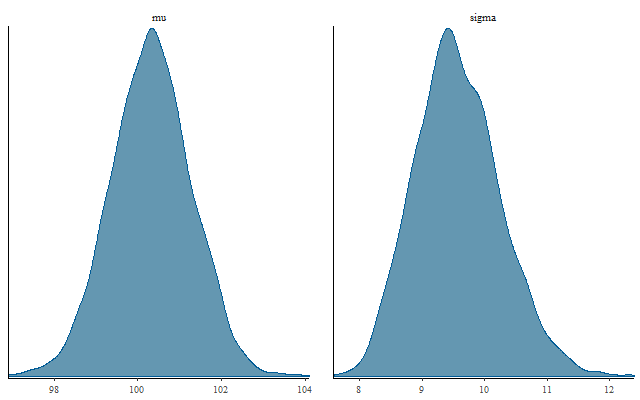
トレースプロット
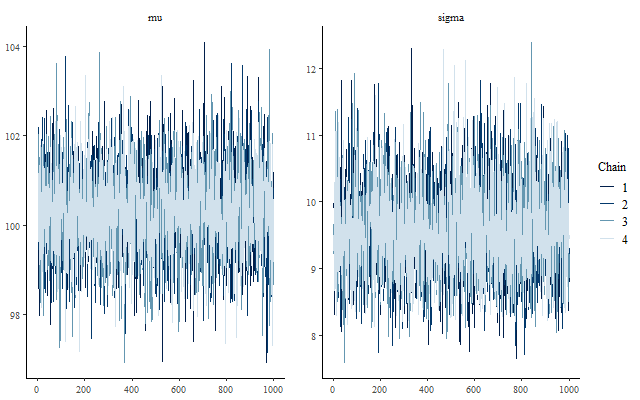
事後分布とトレースプロットを並べて図示
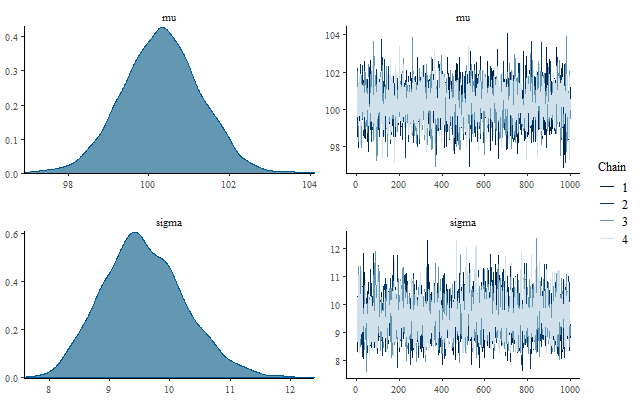
mcmc_combo()メソッドは便利そうですね。
まとめ
Rとstanを使って、正規分布から生成したデータの平均と標準偏差をMCMC法でベイズ推定しました。今後もベイズ統計の勉強を進めていきます。

コメント