diabetesデータセットを使ってLiNGAMで因果探索をやってみたメモです。
LiNGAMチュートリアル
DirectLiNGAM — LiNGAM 1.10.0 documentation
diabetesデータセット

load_diabetes
Gallery examples: Model Complexity Influence Gradient Boosting regression Plot individual and voting regression predictions Model-based and sequential feature s...
内容
準備
# ライブラリインポート
import pandas as pd
import numpy as np
from sklearn.datasets import load_diabetes
from sklearn.preprocessing import StandardScaler
import graphviz
import lingam
from lingam.utils import make_dot, make_prior_knowledge
# データセット読み込み
dataset = load_diabetes(as_frame=True)['data'][['age', 'sex', 'bmi', 'bp']]
dataset['target'] = load_diabetes(as_frame=True)['target']
dataset.head()
age sex bmi bp target
0 0.038076 0.050680 0.061696 0.021872 151.0
1 -0.001882 -0.044642 -0.051474 -0.026328 75.0
2 0.085299 0.050680 0.044451 -0.005671 141.0
3 -0.089063 -0.044642 -0.011595 -0.036656 206.0
4 0.005383 -0.044642 -0.036385 0.021872 135.0DirectLiNGAMで因果探索
# 標準化
scaler = StandardScaler()
Xy = scaler.fit_transform(dataset)
# モデル作成
model = lingam.DirectLiNGAM(random_state=1)
model.fit(Xy)
# 因果の順番
print(model.causal_order_)
# 隣接行列
print(model.adjacency_matrix_)
# 推定した因果モデルにおける、誤差同士の独立性のp値
p_values = model.get_error_independence_p_values(Xy)
print(p_values)[1, 4, 0, 3, 2]
[[0. 0.16595397 0. 0. 0.18074244]
[0. 0. 0. 0. 0. ]
[0. 0. 0. 0.13460604 0.47664595]
[0.23008202 0.1842314 0. 0. 0.39032065]
[0. 0. 0. 0. 0. ]]
[[0. 0.59336161 0.01471721 0.77585358 0.26328001]
[0.59336161 0. 0.00190972 0.44643012 0.73256818]
[0.01471721 0.00190972 0. 0.03910181 0.00928811]
[0.77585358 0.44643012 0.03910181 0. 0.31827615]
[0.26328001 0.73256818 0.00928811 0.31827615 0. ]]
# 因果グラフの描画
# 以下よりgraphvizのexeをインストールする。インストール時にpathに追加する。
# https://graphviz.org/download/
dot = make_dot(model.adjacency_matrix_, labels=dataset.columns.to_list())
dot.format = 'png'
dot.render('DirectLiNGAM')
dot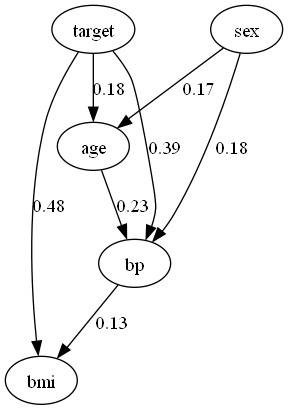
ICA-LiNGAMで因果探索
# 標準化
scaler = StandardScaler()
Xy = scaler.fit_transform(dataset)
# モデル作成
model = lingam.ICALiNGAM(random_state=1)
model.fit(Xy)
# 因果の順番
print(model.causal_order_)
# 隣接行列
print(model.adjacency_matrix_)
# 推定した因果モデルにおける、誤差同士の独立性のp値
p_values = model.get_error_independence_p_values(Xy)
print(p_values)[0, 1, 2, 4, 3]
[[0. 0. 0. 0. 0. ]
[0.1737371 0. 0. 0. 0. ]
[0.11730318 0. 0. 0. 0. ]
[0.2186471 0.17565042 0.16626303 0. 0.29533368]
[0.06725074 0. 0.55633393 0. 0. ]]
[[0.00000000e+00 5.22387240e-28 6.13151850e-03 4.23593615e-01
5.80968024e-02]
[5.22387240e-28 0.00000000e+00 3.45693925e-05 4.28599682e-01
3.50082931e-01]
[6.13151850e-03 3.45693925e-05 0.00000000e+00 2.83284247e-01
1.52685320e-09]
[4.23593615e-01 4.28599682e-01 2.83284247e-01 0.00000000e+00
7.24168274e-01]
[5.80968024e-02 3.50082931e-01 1.52685320e-09 7.24168274e-01
0.00000000e+00]]
dot = make_dot(model.adjacency_matrix_, labels=dataset.columns.to_list())
dot.format = 'png'
dot.render('ICALiNGAM')
dot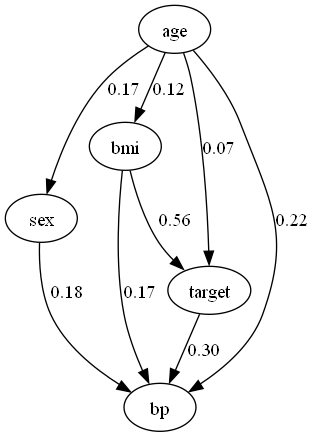
LiNGAMのアルゴリズムによって、推論結果がかなり異なってますね。また、ageとsexに有向辺があることから、データセット自体のバイアスもうかがえます。
時前知識の導入
DirectLiNGAMでは事前知識を導入することができます。
How to use prior knowledge in DirectLiNGAM — LiNGAM 1.10.0 documentation
# 事前知識のグラフを描画する関数
def make_prior_knowledge_graph(prior_knowledge_matrix):
d = graphviz.Digraph(engine='dot')
labels = [f'x{i}' for i in range(prior_knowledge_matrix.shape[0])]
for label in labels:
d.node(label, label)
dirs = np.where(prior_knowledge_matrix > 0)
for to, from_ in zip(dirs[0], dirs[1]):
d.edge(labels[from_], labels[to])
dirs = np.where(prior_knowledge_matrix < 0)
for to, from_ in zip(dirs[0], dirs[1]):
if to != from_:
d.edge(labels[from_], labels[to], style='dashed')
return d
# 事前知識の隣接行列を作成
prior_knowledge = make_prior_knowledge(
n_variables=5,
sink_variables=[4],
)
print(prior_knowledge)
# 事前知識のグラフを描画
dot = make_prior_knowledge_graph(prior_knowledge)
dot.format = 'png'
dot.render('prior_knowledge')
dot[[-1 -1 -1 -1 0]
[-1 -1 -1 -1 0]
[-1 -1 -1 -1 0]
[-1 -1 -1 -1 0]
[-1 -1 -1 -1 -1]]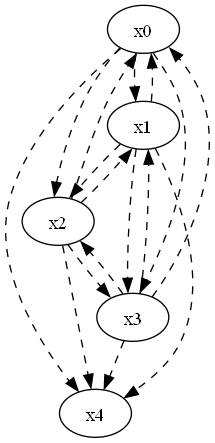
# 標準化
scaler = StandardScaler()
Xy = scaler.fit_transform(dataset)
# モデル作成
model = lingam.DirectLiNGAM(random_state=1, prior_knowledge=prior_knowledge)
model.fit(Xy)
# 因果の順番
print(model.causal_order_)
# 隣接行列
print(model.adjacency_matrix_)
# 推定した因果モデルにおける、誤差同士の独立性のp値
p_values = model.get_error_independence_p_values(Xy)
print(p_values)[1, 0, 2, 3, 4]
[[0. 0.1737371 0. 0. 0. ]
[0. 0. 0. 0. 0. ]
[0.11730318 0. 0. 0. 0. ]
[0.24398632 0.16905852 0.33535276 0. 0. ]
[0. 0. 0.45350717 0.21372887 0. ]]
[[0.00000000e+00 3.60143516e-01 4.92456030e-02 2.44043685e-03
1.81716999e-01]
[3.60143516e-01 0.00000000e+00 2.28878413e-04 4.73044868e-01
1.92837115e-01]
[4.92456030e-02 2.28878413e-04 0.00000000e+00 4.94064284e-02
3.81145593e-07]
[2.44043685e-03 4.73044868e-01 4.94064284e-02 0.00000000e+00
1.48929893e-01]
[1.81716999e-01 1.92837115e-01 3.81145593e-07 1.48929893e-01
0.00000000e+00]]
dot = make_dot(model.adjacency_matrix_, labels=dataset.columns.to_list())
dot.format = 'png'
dot.render('DirectLiNGAM_w/prior_knowledge')
dot事前知識あり
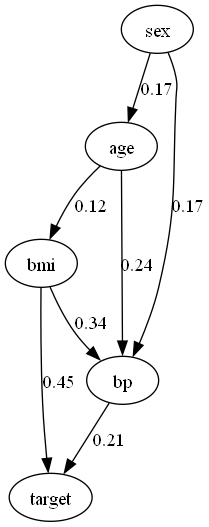
事前知識無し
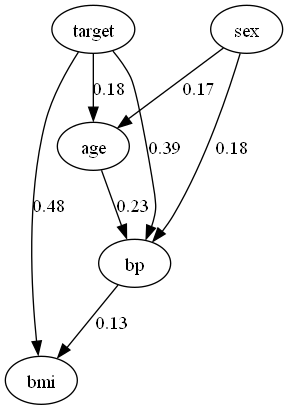
事前知識を導入することで、targetが因果関係の中継地点、又は終点であるという仮定の下で因果探索することができました。
Bootstrap
ブートストラップ法の概要:
- 元データからn個の標本を復元抽出する。このときnは元データの標本数である。
- 統計モデル(モデルパラメータ)を推定する。
- このブートストラップ抽出を何度も(B回)繰り返す。
- こうして計算された「推定量の標本分布」は、本来の標本分布の近似になっている。
Bootstrap — LiNGAM 1.10.0 documentation
from lingam.utils import print_causal_directions, print_dagc
scaler = StandardScaler()
Xy = scaler.fit_transform(dataset)
model = lingam.DirectLiNGAM(random_state=1, prior_knowledge=prior_knowledge)
result = model.bootstrap(Xy, n_sampling=100)
# sklearn.utils.resampleで重複ありのリサンプリングをしている。リサンプル後のサンプル数は元データと同じ。
# bootstrappingの各サンプルにおける、因果方向の有無のカウント結果を取得する
cdc = result.get_causal_direction_counts(n_directions=8, min_causal_effect=0.01, split_by_causal_effect_sign=True)
print_causal_directions(cdc, 100, labels=dataset.columns.to_list())
# カウント結果を確率として、隣接行列の形式で表示
prob = result.get_probabilities(min_causal_effect=0.01)
print(prob)p <--- sex (b>0) (100.0%)
target <--- bmi (b>0) (100.0%)
target <--- bp (b>0) (100.0%)
bp <--- age (b>0) (93.0%)
age <--- sex (b>0) (83.0%)
bp <--- bmi (b>0) (82.0%)
bmi <--- age (b>0) (79.0%)
target <--- sex (b<0) (47.0%)
[[0. 0.83 0.02 0.07 0. ]
[0. 0. 0. 0. 0. ]
[0.79 0.3 0. 0.18 0. ]
[0.93 1. 0.82 0. 0. ]
[0.18 0.47 1. 1. 0. ]]
# bootstrappingの各サンプルにおける、DAG(Directed Acyclic Graphs)のカウント結果を取得する
dagc = result.get_directed_acyclic_graph_counts(n_dags=3, min_causal_effect=0.01, split_by_causal_effect_sign=True)
print_dagc(dagc, 100, labels=dataset.columns.to_list())DAG[0]: 18.0%
age <--- sex (b>0)
bmi <--- age (b>0)
bp <--- age (b>0)
bp <--- sex (b>0)
bp <--- bmi (b>0)
target <--- bmi (b>0)
target <--- bp (b>0)
DAG[1]: 14.0%
age <--- sex (b>0)
bmi <--- age (b>0)
bp <--- age (b>0)
bp <--- sex (b>0)
bp <--- bmi (b>0)
target <--- sex (b<0)
target <--- bmi (b>0)
target <--- bp (b>0)
DAG[2]: 9.0%
age <--- sex (b>0)
bmi <--- age (b>0)
bmi <--- sex (b>0)
bp <--- age (b>0)
bp <--- sex (b>0)
bp <--- bmi (b>0)
target <--- sex (b<0)
target <--- bmi (b>0)
target <--- bp (b>0)
# Total Causal Effects
causal_effects = result.get_total_causal_effects(min_causal_effect=0.01)
# Assign to pandas.DataFrame for pretty display
df = pd.DataFrame(causal_effects)
labels = dataset.columns.to_list()
df['from'] = df['from'].apply(lambda x : labels[x])
df['to'] = df['to'].apply(lambda x : labels[x])
df from to effect probability
0 sex bp 0.222785 1.00
1 bmi target 0.553888 1.00
2 bp target 0.246653 1.00
3 age bp 0.309844 0.93
4 age target 0.166742 0.88
5 age bmi 0.162367 0.84
6 sex age 0.187258 0.83
7 bmi bp 0.351166 0.82
8 sex bmi 0.135094 0.27
9 bp bmi 0.325201 0.18
10 bp age 0.369898 0.07
11 sex target 0.136603 0.04
12 bmi age 0.194196 0.02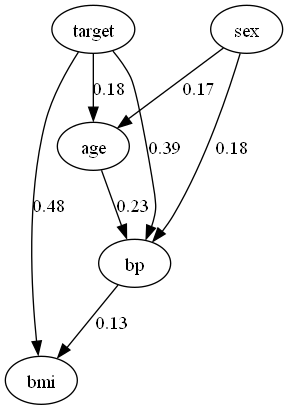

コメント